
DNA-Methyltransferasen
| DNA-Methyltransferasen | ||
|---|---|---|
| Enzymklassifikationen | ||
| EC, Kategorie |
| |
| Reaktionsart | DNA-Methylierung, Übertragung einer Methylgruppe auf den Pyrimidin-Ring des Cytosins in Position 5 durch eine „DNA (cytosine-5-)-methyltransferase“ (EC 2.1.1.37). | |
| Substrat | Cytosin innerhalb von DNA | |
| Produkte | 5-Methylcytosin innerhalb von DNA | |
| EC, Kategorie | | |
| Reaktionsart | DNA-Methylierung, Übertragung einer Methylgruppe auf Position N6 (Aminogruppe am Purin-Ring in Position 6) des Adenins durch eine „Site-specific DNA-methyltransferase (adenine-specific)“ (EC 2.1.1.72). | |
| Substrat | Adenin innerhalb von DNA | |
| Produkte | N6-Methyladenin innerhalb von DNA | |
| EC, Kategorie |
| |
| Reaktionsart | DNA-Methylierung, Übertragung einer Methylgruppe auf Position N4 des Cytosins (auf die Aminogruppe am Pyrimidin-Ring in Position 4) durch eine „Site-specific DNA-methyltransferase (cytosine-N(4)-specific)“ (EC 2.1.1.113). | |
| Substrat | Cytosin innerhalb von DNA | |
| Produkte | N4-Methylcytosin innerhalb von DNA | |
DNA-Methyltransferasen, auch DNA-MTasen genannt, sind Enzyme, die Methylgruppen auf Nukleinbasen der DNA übertragen. Die durch diese Enzyme katalysierte DNA-Methylierung hat eine Vielzahl biologischer Funktionen. In Bakterien werden sie unter anderem dazu genutzt, bakterieneigene DNA zu methylieren, damit beispielsweise Restriktionsenzyme Fremd-DNA und eigene DNA unterscheiden können. Alle bekannten DNA-Methyltransferasen verwenden S-Adenosyl-Methionin (SAM) als Methylgruppendonor.
Klassifizierung
EC-Klassifizierung
Die DNA-MTasen sind nach der Einteilung durch EC-Nummern mit drei Einträgen innerhalb der Methyltransferasen vertreten (EC 2.1.1 – ExPASy[1]), die nach den chemischen Reaktionen, welche sie katalysieren, definiert werden:
- Methylierung von Adenin zu N6-Methyladenin innerhalb von DNA
(EC
 2.1.1.72)
2.1.1.72) - Methylierung von Cytosin zu N4-Methylcytosin innerhalb von DNA
(EC
 2.1.1.113)
2.1.1.113) - Methylierung von Cytosin zu 5-Methylcytosin innerhalb von DNA
(EC
 2.1.1.37)
2.1.1.37)
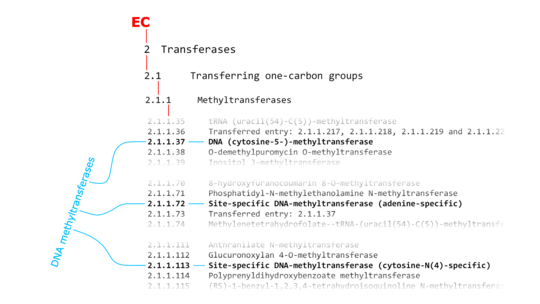
Siehe nebenstehender Text. Innerhalb des EC-Nummern-Systems sind die DNA-Methyltransferasen (im Bild englisch: DNA methyltransferases) keine Enzymklasse (ENZYME class) und kein einzelner Eintrag (ENZYME entry). Die DNA-Methyltransferasen haben drei Einträge (ENZYME entries) innerhalb einer längeren Liste der Methyltransferasen (Methyltransferases; ENZYME class: 2.1.1). Die Methyltransferasen gehören zu den Enzymen, die Gruppen mit einem Kohlenstoff-Atom übertragen (Transferring one-carbon groups; ENZYME class: 2.1), die wiederum zu den Transferasen gehören (Transferases; ENZYME class: 2). Die Einträge für DNA-Methylferasen lauten:
Die Angaben mit dem Wort „ENZYME“ wurden auf dem ExPASy SIB Bioinformatics Resource Portal gefunden (Abruf 17. Oktober 2019, z. B.[1]). |
Einteilung nach dem Methylierungszustand der DNA (Wartung und de novo)
Bei Lebewesen mit ausgeprägter Differenzierung werden die DNA-Methylasen nach ihrem Auftreten in verschiedenen Entwicklungsstadien eingeteilt. Bei Pflanzen und Tieren ist die Differenzierung häufig mit umfassenden Änderungen des Methylierungszustandes der DNA verbunden; das wurde besonders bei verschiedenen Modellorganismen, zum Beispiel bei der Acker-Schmalwand (Arabidopsis thaliana,[2] stellvertretend für die Gefäßpflanzen) und bei der Hausmaus (Mus musculus,[3] stellvertretend für die Säugetiere) untersucht.
De-novo-DNA-Methyltransferasen (englisch de novo DNA methyltransferases) erkennen spezifische Stellen in der DNA, welche es ihnen erlauben, Cytosin de novo zu methylieren. Dies ist bei Säugetieren besonders in der frühen Embryonalentwicklung wichtig, da durch sie ein Methylierungsmuster aufgebaut wird.
DNA-Methyltransferasen zur „Wartung“ (englisch maintenance) fügen Methylgruppen an solchen Stellen der DNA an, an denen an einem DNA-Strang schon eine Methylgruppe vorhanden ist. Dadurch wird das Methylierungsmuster, welches einmal in der Embryonalentwicklung durch die De-novo-DNA-Methyltransferasen aufgebaut wurde, erhalten.
Literatur
- W. A. Loenen, A. S. Daniel, H. D. Braymer, N. E. Murray: Organization and sequence of the hsd genes of Escherichia coli K-12. In: J. Mol. Biol. 198 (2), November 1987,
S. 159–170.
 doi:10.1016/0022-2836(87)90303-2.
doi:10.1016/0022-2836(87)90303-2.
 PMID 3323532.
PMID 3323532. - K. E. Narva, J. L. Van Etten, B. E. Slatko, J. S. Benner: The amino acid sequence of the eukaryotic DNA [N6-adenine]methyltransferase, M.CviBIII, has regions of similarity with the
prokaryotic isoschizomer M.TaqI and other DNA [N6-adenine] methyltransferases. In: Gene. 74 (1), Dezember 1988, S. 253–259.
 doi:10.1016/0378-1119(88)90298-3.
doi:10.1016/0378-1119(88)90298-3.
 PMID 3248728.
PMID 3248728. - R. Lauster: Evolution of type II DNA methyltransferases. A gene duplication model. In: J. Mol. Biol. 206 (2), März 1989, S. 313–321.
 doi:10.1016/0022-2836(89)90481-6.
doi:10.1016/0022-2836(89)90481-6.
 PMID 2541254.
PMID 2541254. - A. Timinskas, V. Butkus, A. Janulaitis: Sequence motifs characteristic for DNA [cytosine-N4] and DNA [adenine-N6] methyltransferases. Classification of all DNA
methyltransferases. In: Gene. 157 (1–2), Mai 1995, S. 3–11.
 doi:10.1016/0378-1119(94)00783-O.
doi:10.1016/0378-1119(94)00783-O.
 PMID 7607512.
PMID 7607512. - J. Labahn, J. Granzin, G. Schluckebier, D. P. Robinson, W. E. Jack, I. Schildkraut, W. Saenger: Three-dimensional structure of the adenine-specific DNA methyltransferase M.Taq I in
complex with the cofactor S-adenosylmethionine. In: Proc. Natl. Acad. Sci. U.S.A. 91 (23), November 1994, S. 10957–10961.
 doi:10.1073/pnas.91.23.10957.
doi:10.1073/pnas.91.23.10957.
 PMC 45145 (freier Volltext).
PMC 45145 (freier Volltext).
 PMID 7971991.
PMID 7971991. - J. E. Kelleher, A. S. Daniel, N. E. Murray: Mutations that confer de novo activity upon a maintenance methyltransferase. In: Journal of Molecular Biology. 221 (2), 1991,
S. 431–440.
 doi:10.1016/0022-2836(91)80064-2.
doi:10.1016/0022-2836(91)80064-2.
 PMID 1833555.
PMID 1833555. - J. Pósfai, A. S. Bhagwat, R. J. Roberts: Sequence motifs specific for cytosine methyltransferases. In: Gene. 74 (1), Dezember 1988, S. 261–265.
 doi:10.1016/0378-1119(88)90299-5.
doi:10.1016/0378-1119(88)90299-5.
 PMID 3248729.
PMID 3248729. - S. Kumar, X. Cheng, S. Klimasauskas, S. Mi, J. Posfai, R. J. Roberts, G. G. Wilson: The DNA (cytosine-5) methyltransferases. In: Nucleic Acids Res. 22 (1), Januar 1994,
S. 1–10.
 doi:10.1093/nar/22.1.1.
doi:10.1093/nar/22.1.1.
 PMC 307737 (freier Volltext).
PMC 307737 (freier Volltext).
 PMID 8127644.
PMID 8127644. - R. Lauster, T. A. Trautner, M. Noyer-Weidner: Cytosine-specific type II DNA methyltransferases. A conserved enzyme core with variable target-recognizing domains. In: J. Mol.
Biol. 206 (2), März 1989, S. 305–312.
 doi:10.1016/0022-2836(89)90480-4.
doi:10.1016/0022-2836(89)90480-4.
 PMID 2716049.
PMID 2716049. - X. Cheng: DNA modification by methyltransferases. In: Curr. Opin. Struct. Biol. 5 (1), Februar 1995, S. 4–10.
 doi:10.1016/0959-440X(95)80003-J.
doi:10.1016/0959-440X(95)80003-J.
 PMID 7773746.
PMID 7773746.
Einzelnachweise
- ↑ Hochspringen nach: a b
 Enymklasse 2.1.1 (enzyme.expasy.org/EC/2.1.1.-). In:
ExPASy SIB Bioinformatics Resource Portal. SIB Swiss Institute of Bioinformatics,
(englisch, Release 16-Oct-19).
Enymklasse 2.1.1 (enzyme.expasy.org/EC/2.1.1.-). In:
ExPASy SIB Bioinformatics Resource Portal. SIB Swiss Institute of Bioinformatics,
(englisch, Release 16-Oct-19).
- ↑ Marc W. Schmid, Christian Heichinger, Diana Coman Schmid, Daniela Guthörl, Valeria Gagliardini:
Contribution of epigenetic variation to adaptation in Arabidopsis. In: Nature Communications.
Band 9,
Nr. 1, 25. Oktober 2018,
ISSN 2041-1723, S. 4446,
doi:
 10.1038/s41467-018-06932-5,
10.1038/s41467-018-06932-5,
 PMID 30361538,
PMID 30361538,
 PMC 6202389 (freier Volltext).
PMC 6202389 (freier Volltext).
- ↑ Jennifer M. SanMiguel, Marisa S. Bartolomei: DNA methylation dynamics of
genomic imprinting in mouse development. In: Biology of Reproduction.
Band 99,
Nr. 1, 1. Juli 2018,
ISSN 1529-7268, S. 252–262,
doi:
 10.1093/biolre/ioy036,
10.1093/biolre/ioy036,
 PMID 29462489,
PMID 29462489,
 PMC 6044325 (freier Volltext).
PMC 6044325 (freier Volltext).


© biancahoegel.de
Datum der letzten Änderung: Jena, den: 24.11. 2025