
RNA-Polymerasen
| RNA-Polymerasen | ||
|---|---|---|
| ||
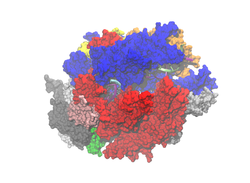
| Oberflächenmodell des RNA-Polymerase-II-Komplexes der Bäckerhefe (jede der 10 Untereinheiten unterschiedlich gefärbt), nach PDB 3G1G; RNA (links) und DNA (links+rechts) als Cartoon | ||
| Enzymklassifikationen | ||
| EC, Kategorie | | |
| Substrat | Nucleosidtriphosphat + RNAn | |
| Produkte | Diphosphat + RNAn+1 | |
| EC, Kategorie | | |
| Reaktionsart | Addition einer Ribonukleinsäure | |
| Substrat | Nucleosidtriphosphat + RNAn | |
| Produkte | Diphosphat + RNAn+1 | |
RNA-Polymerasen sind in der Regel Proteinkomplexe, die als Enzyme beim Aufbau des strangförmigen Polymers einer Ribonukleinsäure (RNA) aus ihren Grundbausteinen (Ribonukleotide) mitwirken. Diese aus mehreren Untereinheiten zusammengesetzten Polymerasen werden danach unterschieden, ob sie für ihre Wirkung bei der Synthese eines RNA-Stranges von der Vorlage einer Matrize aus DNA oder RNA abhängig sind oder nicht. DNA-abhängige RNA-Polymerasen spielen eine wesentliche Rolle bei der Transkription und kommen daher in allen Lebewesen vor.
Für die RNA-Welt-Hypothese Hypothese von Bedeutung sind Ribozyme (d. h. enzymatisch wirksame RNAs), die als RNA-Polymerasen wirken.
Bakterielle DNA-abhängige RNA-Polymerasen
Bei Bakterien gibt es eine DNA-abhängige RNA-Polymerase, die an der Expression aller Gene beteiligt ist. Die prokaryotische RNA-Polymerase besteht aus den Untereinheiten α, β, β' und dem σ-Faktor, wobei die α-Untereinheit zweimal vorliegt, die anderen je einmal. Das α2-Dimer ist für den Zusammenbau (Assembly) der anderen Untereinheiten notwendig und bindet regulatorische Proteine, die β-Untereinheit bindet die Nucleosid-5'-triphosphate und katalysiert die Bildung der Phosphodiesterbindung, die β'-Untereinheit hat DNA-bindende Funktion. Der σ-Faktor schließlich erkennt und bindet an den Promotor. Unter Stressbedingungen werden andere σ-Faktoren eingesetzt; diese können an die speziellen Promotoren besonderer Gene binden. Bei Escherichia coli etwa wird der Hitzeschockgenpromotor durch die normale RNA-Polymerase mit der σ70-Untereinheit nicht erkannt, jedoch durch eine σ32-RNA-Polymerase, die bei einem Hitzeschock aktiv ist.
Für die Synthese der RNA-Primer der Replikation haben Bakterien eine zusätzliche DNA-abhängige RNA-Polymerase, die Primase DnaG.[1]
Eukaryotische DNA-abhängige RNA-Polymerasen
Bei Eukaryoten gibt es mehrere verschiedene Formen DNA-abhängige RNA-Polymerasen im Zellkern:
- die RNA-Polymerase I katalysiert die Bildung von prä-rRNA 45S (wird prozessiert zu 18S, 5.8S und 28S) im Nucleolus;
- die RNA-Polymerase II katalysiert die Bildung von prä-mRNA, snoRNAs (small nucleolar RNAs) und mancher snRNAs (small nuclear RNA) sowie siRNA und miRNA;[2]
- die RNA-Polymerase III katalysiert die Bildung von tRNA, 5S-rRNA, 7SL-RNA, einiger snRNAs und anderer kleiner RNAs;
- die RNA-Polymerase IV katalysiert die Bildung von siRNA und wird dabei von der RNA-Polymerase V unterstützt.
Die RNA-Polymerasen II und III werden durch das in verschiedenen Pilzen vorkommende α-Amanitin gehemmt.[3]
Virale RNA-abhängige RNA-Polymerasen
Daneben existieren auch RNA-abhängige RNA-Polymerasen, die bei der Vermehrung des Erbguts von RNA-Viren helfen.
Unabhängige RNA-Polymerasen
Ein Beispiel für eine unabhängige RNA-Polymerase ist die Poly(A)-Polymerase, die für die Polyadenylierung der mRNA sorgt.
Biochemische Aspekte
RNA-Polymerasen verfügen über einen einfachen Mechanismus zur Fehlererkennung: Wenn sich an eine Base der DNA ein unpassendes RNA-Nukleotid anlagert, so verbleibt die RNA-Polymerase länger an der entsprechenden DNA-Stelle. Dadurch wächst die Wahrscheinlichkeit, dass sich das schlecht gepaarte Ribonukleotid wieder von der DNA entfernt. Insgesamt wird durch diesen Mechanismus eine Genauigkeit von einem Fehler auf 10.000 Basenpaarungen erreicht. Dies entspricht etwa einem Fehler pro synthetisiertem RNA-Molekül.
Die RNA-Synthese erfolgt wie die der DNA-Replikation in 5'→3'-Richtung, womit das 3'-Ende des komplementären DNA-Strangabschnitts (bei der Replikation als „Leitstrang“ bezeichnet) dem 5'-Ende der mRNA sowie dem N-terminalen Ende des bei der Translation entstehenden Proteins (Colinearität) entspricht und umgekehrt, das 5'-Ende des abgelesenen DNA-Strangabschnitts also dem 3'-Ende der entstandenen mRNA sowie dem C-Terminus des Proteins entspricht.
Anders gesagt, wird die RNA-Sequenz damit entlang des komplementären DNA-„Leitstrangs“ als Matrize von dessen 3'- zu seinem 5'-Ende gelesen und dabei selbst in 5´→3´-Richtung synthetisiert, wie dann auch als mRNA in 5´→3´-Richtung vom Ribosom abgelesen und in das Protein übersetzt (N-terminal → C-terminal), was es z. B. Bakterien ermöglicht, an einer noch gar nicht komplett fertiggestellten mRNA an deren 5'-Ende bereits mit der Proteinsynthese zu beginnen.
Im Unterschied zur Replikation der DNA und deren DNA-Polymerasen jedoch benötigen RNA-Polymerasen hierzu kein freies 3'-OH und somit auch keine Primer.
Die RNA-Polymerasen sind komplex aufgebaut. Bei der Hefe (Saccharomyces) sind zehn verschiedene Polypeptid-Ketten, deren Molekularmasse zwischen 7.700 und 140.000 Dalton liegen, Magnesium, Zink sowie zwei DNA-Ketten beteiligt. Insgesamt besteht diese RNA-Polymerase aus über 28.000 Atomen.
Bei Escherichia coli wird der RNA-Strang durch die RNA-Polymerase mit einer Rate von ca. 50 Nukleotiden pro Sekunde (17 nm/s) verlängert.
Für die Aufklärung des Mechanismus der Transkription mittels der RNA-Polymerase erhielt der US-amerikanische Chemiker Roger D. Kornberg 2006 den Nobelpreis für Chemie.
RNA-abhängige RNA-Polymerase-Ribozyme
Die Entdeckung von RNA-Polymerase Ribozymen (d. h. als RNA-Polymerase katalytisch wirksamer RNA) mit ausreichend hoher Wiedergabetreue durch Papastavrou et al. am Salk Institute (2024) ist für die RNA-Welt-Hypothese von großer Bedeutung, da sie Voraussetzung für eine RNA-katalysierte Evolution der Ribozyme ist.[4]
Einzelnachweise
- ↑ Stefan Ilic, Shira Cohen, Meenakshi Singh, Benjamin Tam, Adi Dayan:
DnaG Primase—A Target for the Development of Novel Antibacterial Agents. In: Antibiotics.
Band 7,
Nr. 3, 13. August 2018, ISSN 2079-6382,
doi:
 10.3390/antibiotics7030072,
10.3390/antibiotics7030072,
 PMID 30104489,
PMID 30104489,
 PMC 6163395 (freier Volltext).
PMC 6163395 (freier Volltext).
- ↑ Alberts, et al. Fifth Edition P. 340
- ↑ E. Brändle, K. Zetsche: Zur Lokalisation der α-Amanitin sensitiven RNA-Polymerase
in Zellkernen von Acetabularia. In: Planta.
Band 111,
Nr. 3, 1. September 1973, ISSN 1432-2048,
S. 209–217,
doi:
 10.1007/BF00385105.
10.1007/BF00385105.
- ↑
Nikolaos Papastavrou, David P. Horning, Gerald F. Joyce: RNA-catalyzed evolution of catalytic RNA. In: PNAS, Band 121, Nr. 11, 4. März 2024, S. e23215921;
 doi:10.1073/pnas.2321592121
(englisch). Dazu:
doi:10.1073/pnas.2321592121
(englisch). Dazu:
- Claudia Krapp:
 Neuer Einblick in die „RNA-Welt“.
Wie sich RNA-Enzyme auf der Urerde evolutionär optimieren konnten. Auf: scinexx.de vom 5. März 2024.
Neuer Einblick in die „RNA-Welt“.
Wie sich RNA-Enzyme auf der Urerde evolutionär optimieren konnten. Auf: scinexx.de vom 5. März 2024. -
 Unlocking the Secrets of Life With RNA’s Ancient Code.
Auf: SciTechDaily vom 1. April 2024. Quelle: Salk Institute.
Unlocking the Secrets of Life With RNA’s Ancient Code.
Auf: SciTechDaily vom 1. April 2024. Quelle: Salk Institute.
- Claudia Krapp:


© biancahoegel.de
Datum der letzten Änderung: Jena, den: 14.09. 2025